13.3 R como una plataforma SIG
13.3.1 Introducción
El análisis espacial y espacio-temporal es una extensión natural a las capacidades de R. Existen múltiples paquetes para manipular y analizar datos espacio-temporales en R. En esta clase vamos a dar una introducción a esto.
Primero que nada establezcamos el directorio de trabajo
13.3.2 Matrices y data frames
Recordando, una matriz es un arreglo bidimensional con una cierta cantidad de renglones y de columnas, por ejemplo un cuadrado de \(3*3\) que contenga los números del 1 al 9
matriz <- matrix(seq(1,9),nrow=3,ncol=3)
matriz## [,1] [,2] [,3]
## [1,] 1 4 7
## [2,] 2 5 8
## [3,] 3 6 9Un data.frame es también un arreglo con la diferencia de que puede contener más de un tipo de datos. Esto es, puede alojar texto, números y valores booleanos simultáneamente.
numeros <- c(1,2,3)
texto <- c("hola","como","estas")
booleanos <- c(TRUE,FALSE,TRUE)
data_frame <- data.frame(numeros,texto,booleanos)
data_frame## numeros texto booleanos
## 1 1 hola TRUE
## 2 2 como FALSE
## 3 3 estas TRUEAmbos tienen asociadas coordenadas de la forma \((renglón,columna)\) que comienzan de arriba a abajo y de izquierda a derecha. Estas permiten recuperar los valores que guardan, por ejemplo la entrada \((renglón,columna)=(2,1)\) para la matriz y el data.frame que definimos antes corresponden a:
matriz[2,1]## [1] 2data_frame[2,1]## [1] 2También se pueden hacer búsquedas condicionales sobre estos objetos, esto es escribir en código peticiones como: dime qué valores de la primera columna de la matriz son mayores a 1.
Cuando se deja vacía una dimensión, R entiende que le estás pidiendo que incluya todos los valores existentes en ella. Por tanto, la pregunta ¿qué valores de la columna 1 de la matriz son mayores a 1? se expresa de la siguiente manera:
condicion <- matriz[,1]>1
condicion## [1] FALSE TRUE TRUELa primer columna está compuesta por los valores 1 (que claramente no es mayor a 1), 2 y 3 por lo tanto el resultado es FALSE, TRUE, TRUE.
La misma manera de hacerle “preguntas” a una matriz se puede lograr con un data.frame
condicion <- data_frame[,1]>1
condicion## [1] FALSE TRUE TRUEAlgunas preguntas, por la naturaleza de los datos no tienen sentido. Por ejemplo de las palabras “hola”, “como”, “estas” ¿cuáles son mayores a 1?
13.3.3 Rasters, primeros pasos
Un raster es simplemente una matriz de números qué tiene la intención de usarse como base para generar imágenes. A estos se les puede asociar una estructura espacial.
En R, existe el paquete “raster” que está ampliamente desarrollado y se recomienda como espina dorsal para cualquier análisis que incorpore imágenes de satélite.
La mayoría de las imágenes de satélite se pueden cargar usando la función raster(). Esta misma permite insertar una matriz en un objeto raster de R.
library("raster")## Loading required package: spmatriz_raster <- raster(matriz)
matriz_raster## class : RasterLayer
## dimensions : 3, 3, 9 (nrow, ncol, ncell)
## resolution : 0.3333333, 0.3333333 (x, y)
## extent : 0, 1, 0, 1 (xmin, xmax, ymin, ymax)
## coord. ref. : NA
## data source : in memory
## names : layer
## values : 1, 9 (min, max)Como podrá notarse, el objeto matriz_raster es ya un raster. Si bien, uno poco interesante y sin proyección.
Se mencionó que los rasters están pensados para ser la base de imágenes. El paquete “raster” contiene funcionalidades básicas de visualización de objetos raster.
plot(matriz_raster)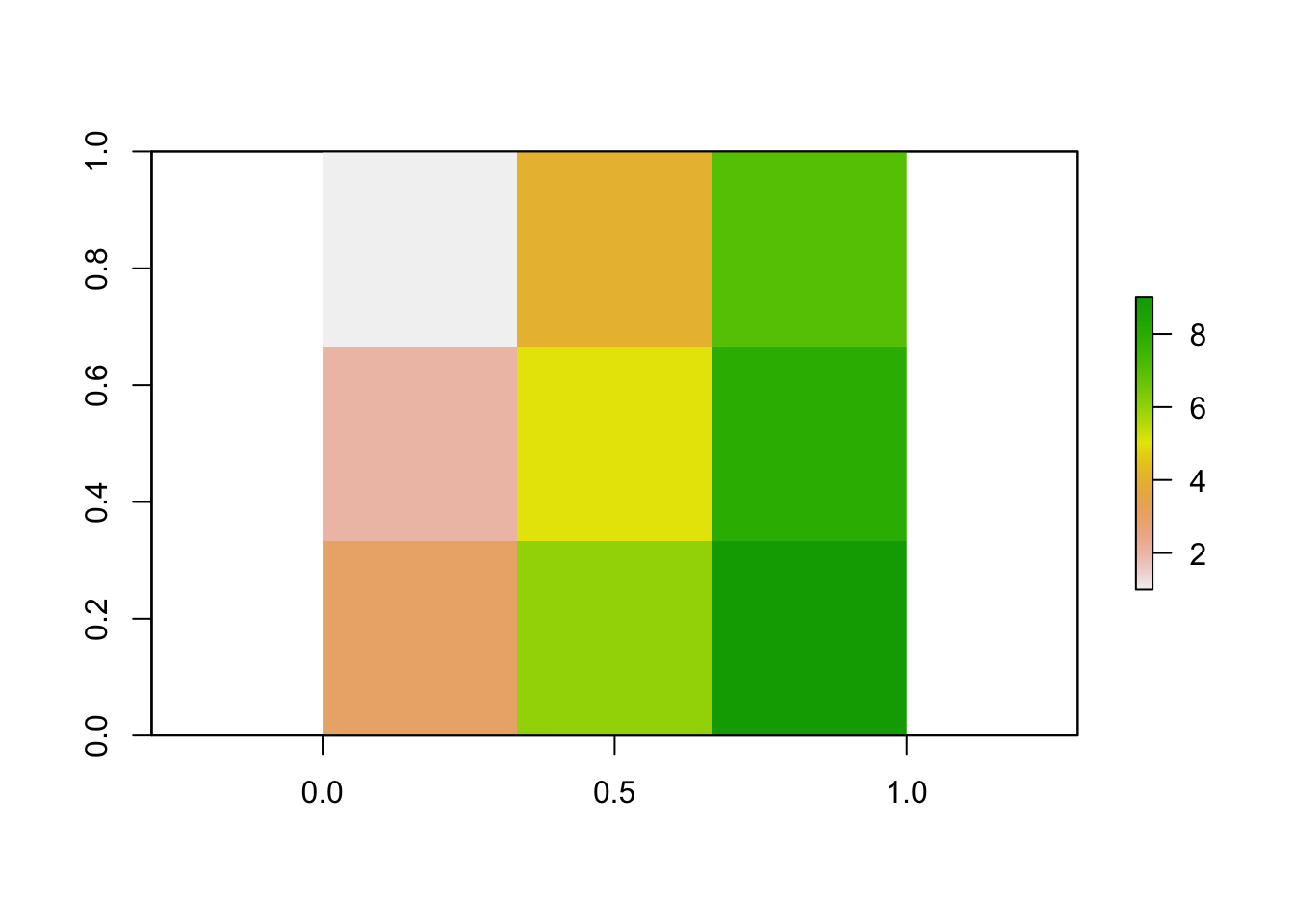
Es muy importante observar que un objeto raster tiene asociados dos conjuntos de coordenadas. El objeto matriz_raster anterior se generó a partir de una matriz de dimensión \(3*3\) por lo que está conformado por 9 píxeles que se ubican a través de su (renglón,columna). Por otro lado, define un espacio abstracto continuo localizado en el extent: 0, 1, 0, 1 (xmin, xmax, ymin, ymax). Por tanto la resolución (tamaño de cada píxel) del raster es de \(0.333*0.333\).
En este contexto, los puntos son objetos espaciales sin área. Por lo que uno se puede localizar en cualquier lugar del espacio que definimos, por ejemplo en las coordenadas \((0.25,0.75)\)
plot(matriz_raster)
points(0.25,0.75,pch=21,bg="red",cex=2)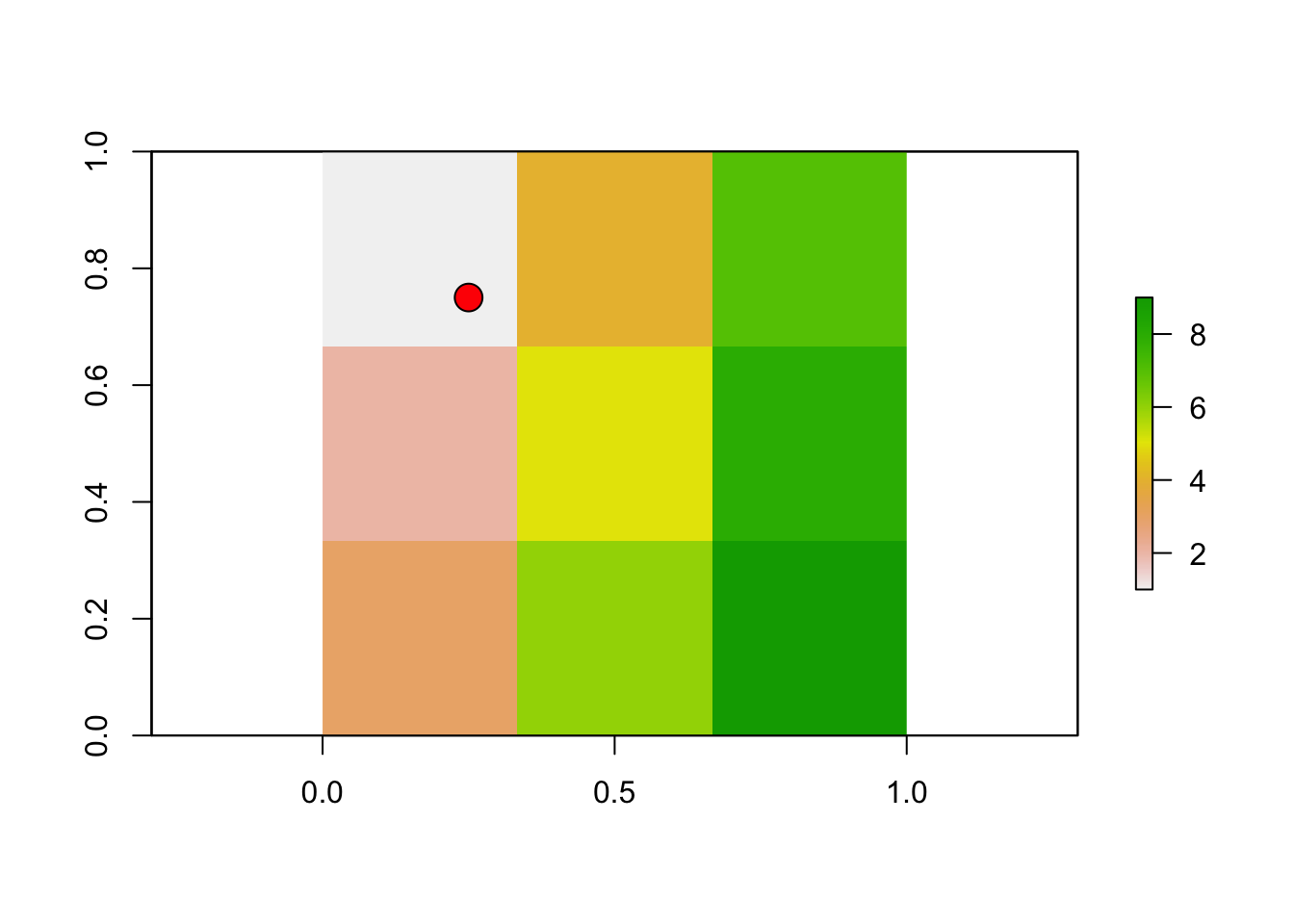
Utilizando el paquete “rasterVis” que es uno cuya intención primordial es visualizar rasters y sus análisis podemos visualizar este raster muy simple con los valores correspondientes a cada pixel. Esto permitirá observar el resultado de los siguientes procesos que llevaremos a cabo.
library("ggplot2")
library("rasterVis")## Loading required package: lattice## Loading required package: latticeExtra## Loading required package: RColorBrewer##
## Attaching package: 'latticeExtra'## The following object is masked from 'package:ggplot2':
##
## layergplot(matriz_raster) + geom_tile(aes(fill=values(matriz_raster))) +
scale_colour_brewer(palette="Blues") +
coord_equal() +
geom_text(aes(label=sprintf("%02.0f",values(matriz_raster))),color="white",size=5)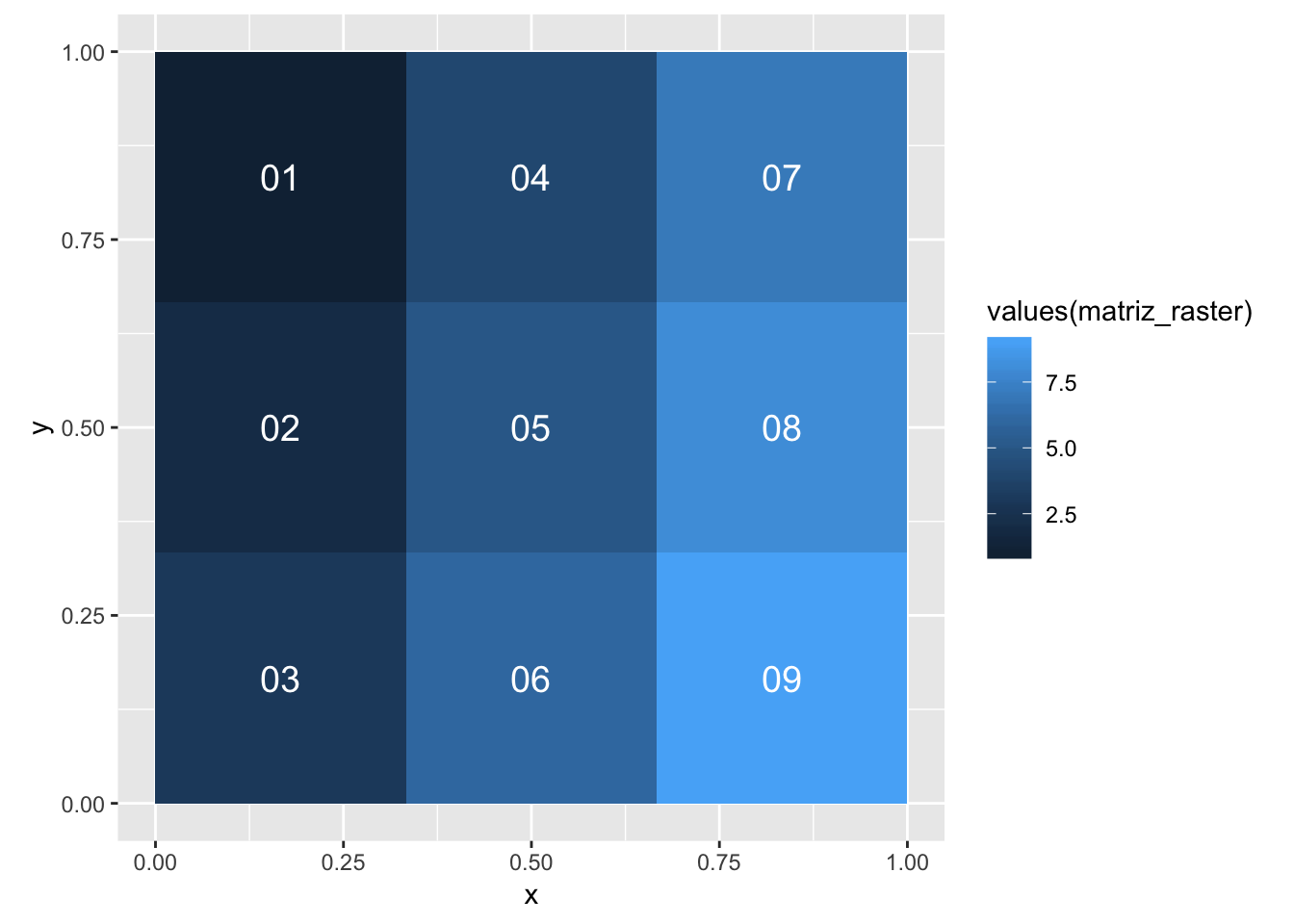
Análogamente a como sucedía con la matriz, podemos recuperar el valor del raster en la entrada \((renglón,columna)=(2,1)\)
matriz_raster[2,1]##
## 2Un raster también es entendido en R como un vector de arriba a abajo y de izquierda a derecha. En este caso el arreglo tiene longitud 9 y la entrada \((renglón,columna)=(2,1)\) es igual a la entrada \([4]\).
matriz_raster[4]##
## 2Lo anterior en combinación con expresiones condicionales nos permite hacer búsquedas y manipular rasters.
Podemos multiplicar el raster en su totalidad por 2 y restarle la constante 1.
matriz_raster_por2_menos1 <- matriz_raster * 2 - 1
gplot(matriz_raster_por2_menos1) + geom_tile(aes(fill=values(matriz_raster_por2_menos1))) +
scale_colour_brewer(palette="Blues") +
coord_equal() +
geom_text(aes(label=sprintf("%02.0f",values(matriz_raster_por2_menos1))),color="white",size=5)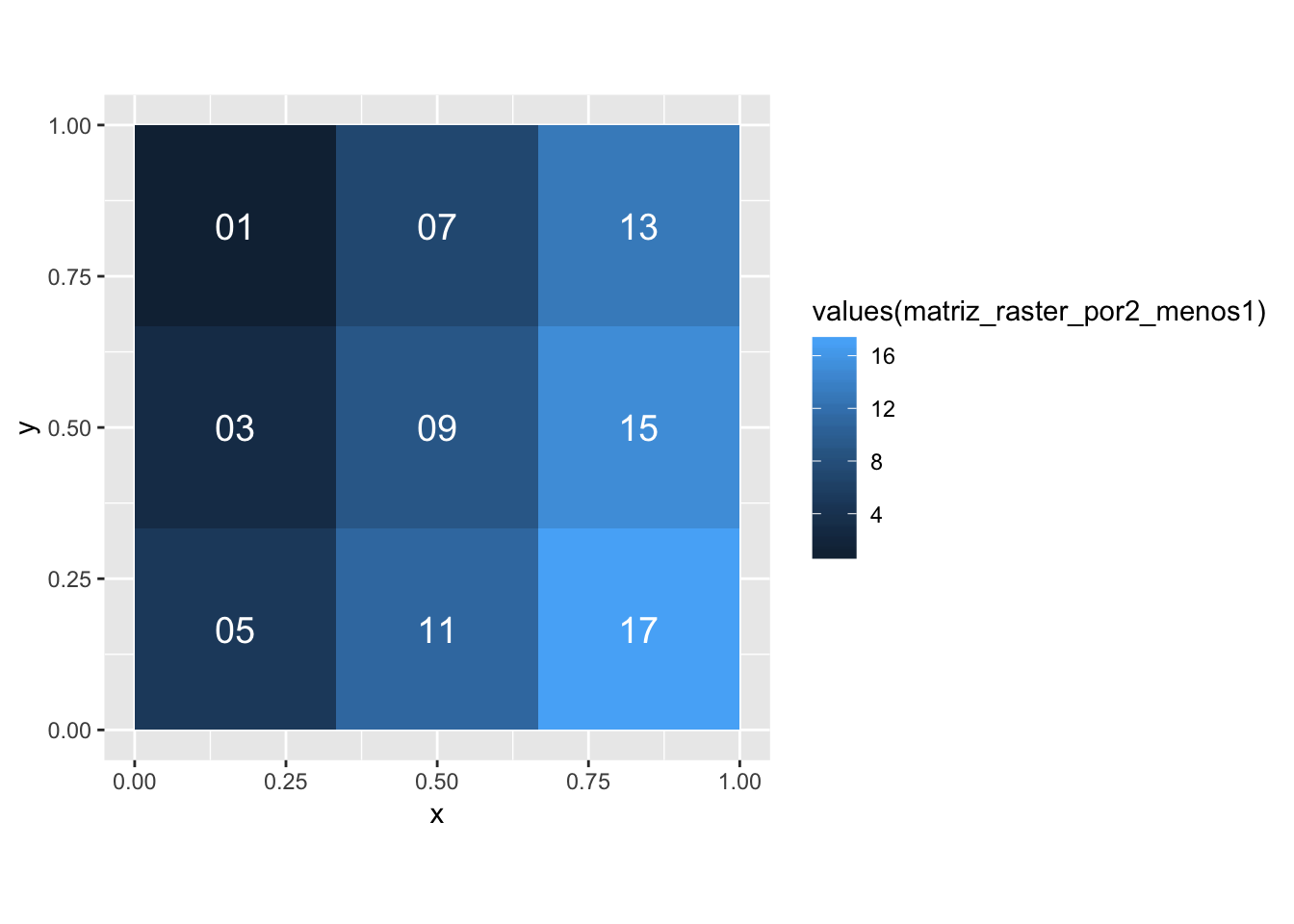
Podemos reemplazar todos los píxeles del raster que cumplan la condición de ser mayores a 4 por 999. Esto se logra expresando la condición de manera análoga a como lo hicimos para matrices. Como un raster se puede ver como un vector, basta con meter la condición en una única dimensión
# reemplazar matriz_raster donde matriz_raster mayor a 4 por 999
matriz_raster[matriz_raster>4] <- 999
gplot(matriz_raster) + geom_tile(aes(fill=values(matriz_raster))) +
scale_colour_brewer(palette="Blues") +
coord_equal() +
geom_text(aes(label=sprintf("%02.0f",values(matriz_raster))),color="white",size=5)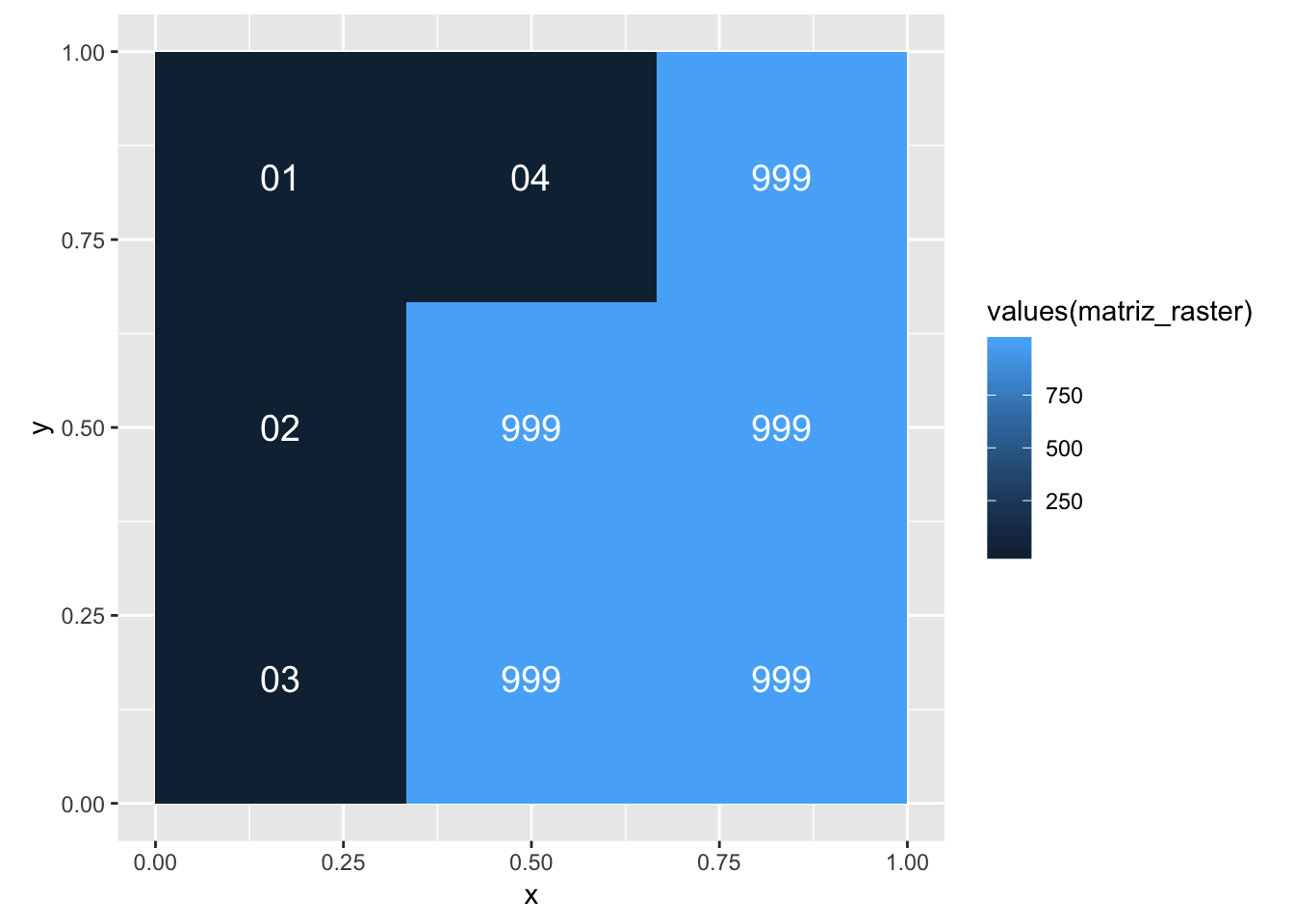
Hay funciones básicas en el paquete raster que son útiles en mútiples situaciones:
cellStats() calcula un estadístico definido por el usuario sobre un raster de entrada, por ejemplo se puede usar para obtener el promedio de matriz_raster
cellStats(matriz_raster,stat="mean")## [1] 556.111113.3.4 Análisis de Rasters usando R
Como se indicó se puede cargar prácticamente cualquier raster utilizando la función raster(). Muchas imágenes satelitales son en realidad multiespectrales, esto quiere decir que en vez de ser una sola matriz son varias apiladas. Por ejemplo las imágenes RapidEye son imágenes constan de 5 bandas (green,blue,red, red edge, near infrared). Estas imágenes en particular se deben cargar con la función brick() o stack(). Carguemos un recorte de imagen RapidEye localizada en el estado de Chiapas. Si se llama al objeto se desplegarán los metadatos del mismo:
chiapas1 <- brick("./data/1crop.tif")
chiapas1## class : RasterBrick
## dimensions : 964, 1056, 1017984, 5 (nrow, ncol, ncell, nlayers)
## resolution : 5, 5 (x, y)
## extent : 722995, 728275, 1789575, 1794395 (xmin, xmax, ymin, ymax)
## coord. ref. : +proj=utm +zone=15 +datum=WGS84 +units=m +no_defs +ellps=WGS84 +towgs84=0,0,0
## data source : /Users/jequihua/Documents/repositories/big-data-ambiental/data/1crop.tif
## names : X1crop.1, X1crop.2, X1crop.3, X1crop.4, X1crop.5
## min values : 3706, 2469, 1208, 974, 722
## max values : 13994, 13601, 12661, 11159, 15283# visualizar las bandas RGB
plotRGB(chiapas1,r=3,g=2,b=1)
Utilizando la función subset() podemos obtener una o más bandas de una imagen multiespectral. Por ejemplo podemos extraer las bandas red (VIS) y near infrared (NIR).
VIS <- subset(chiapas1,subset=3)
NIR <- subset(chiapas1,subset=5)
par(mfrow=c(1,2))
plot(VIS,main="VIS")
plot(NIR,main="NIR")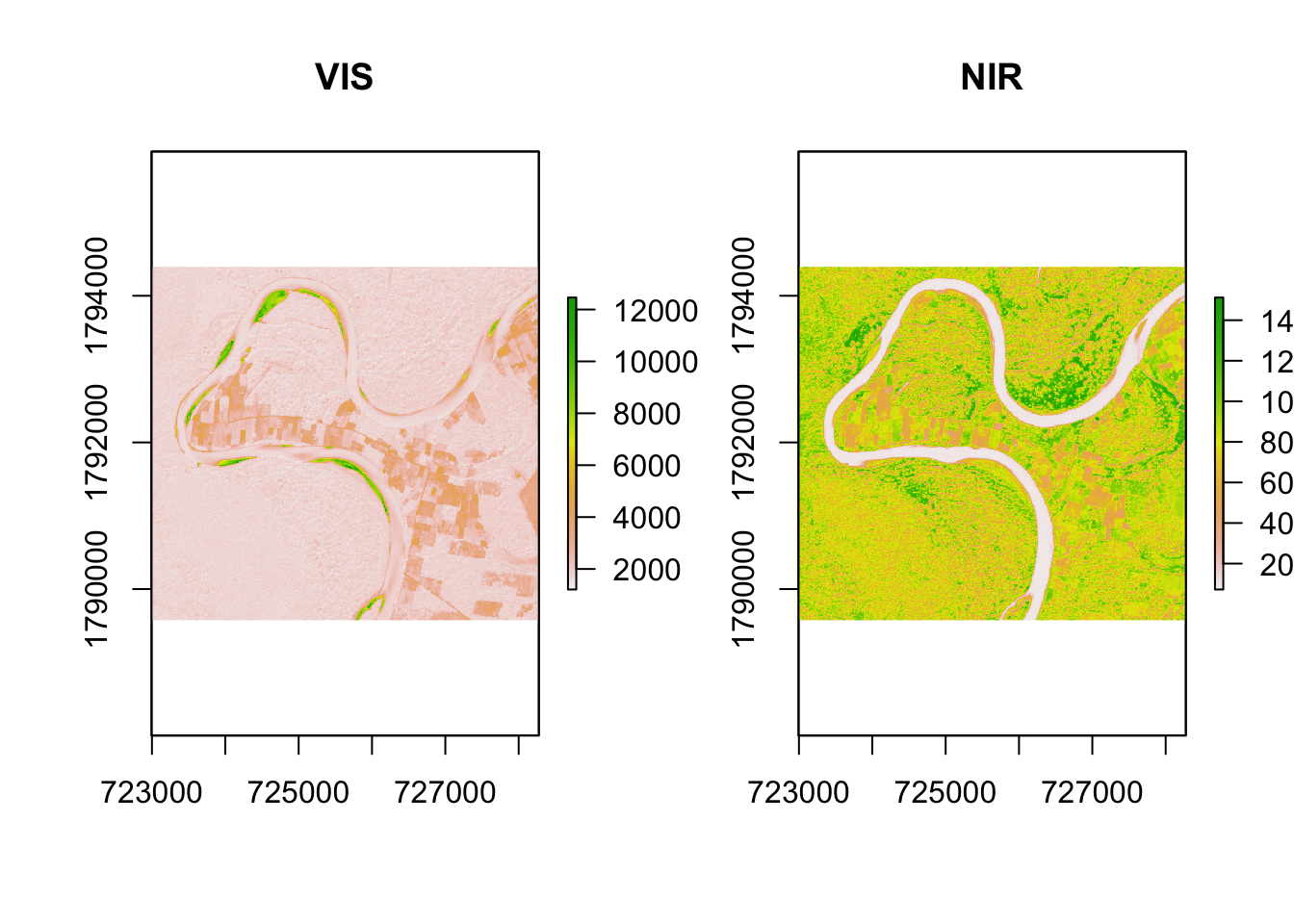
Utilizando los mecanismos de algebra de mapas descritos en la sección anterior podemos trivialmente calcular el NDVI de esta imagen.
\[ \begin{aligned} NDVI := \frac{NIR-VIS}{NIR+VIS} \end{aligned} \]
chiapas1_ndvi <- (NIR-VIS)/(NIR+VIS)
plot(chiapas1_ndvi,main="NDVI")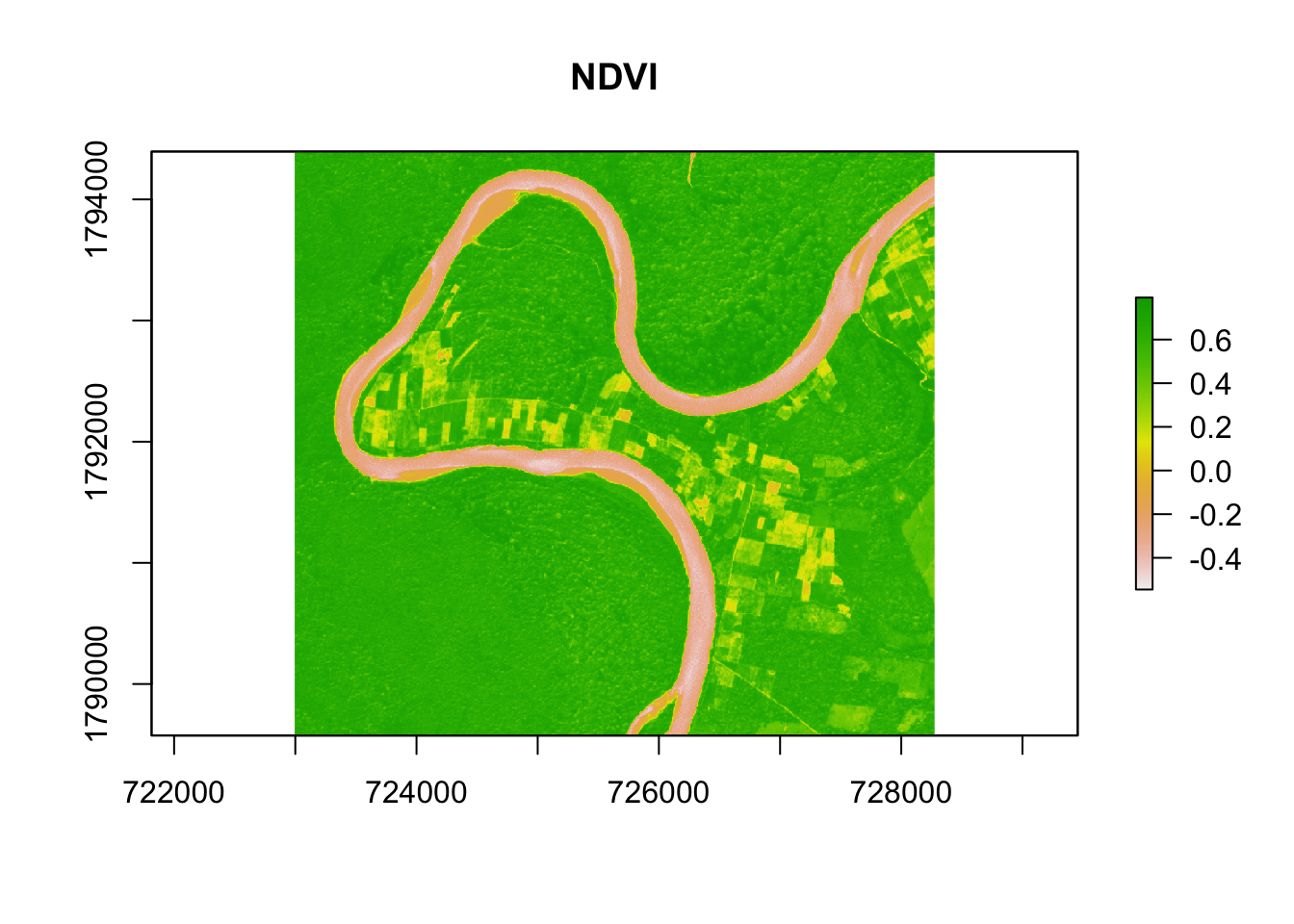
Utilizando algebra de mapas y la función cellStats() podemos estandarizar esta imagen. Le restaremos su media y dividiremos esto sobre la desviación estándar de la misma.
chiapas1_ndvi_st <- (chiapas1_ndvi-cellStats(chiapas1_ndvi,stat="mean"))/(cellStats(chiapas1_ndvi,stat="sd"))
plot(chiapas1_ndvi_st,main="NDVI estandarizado")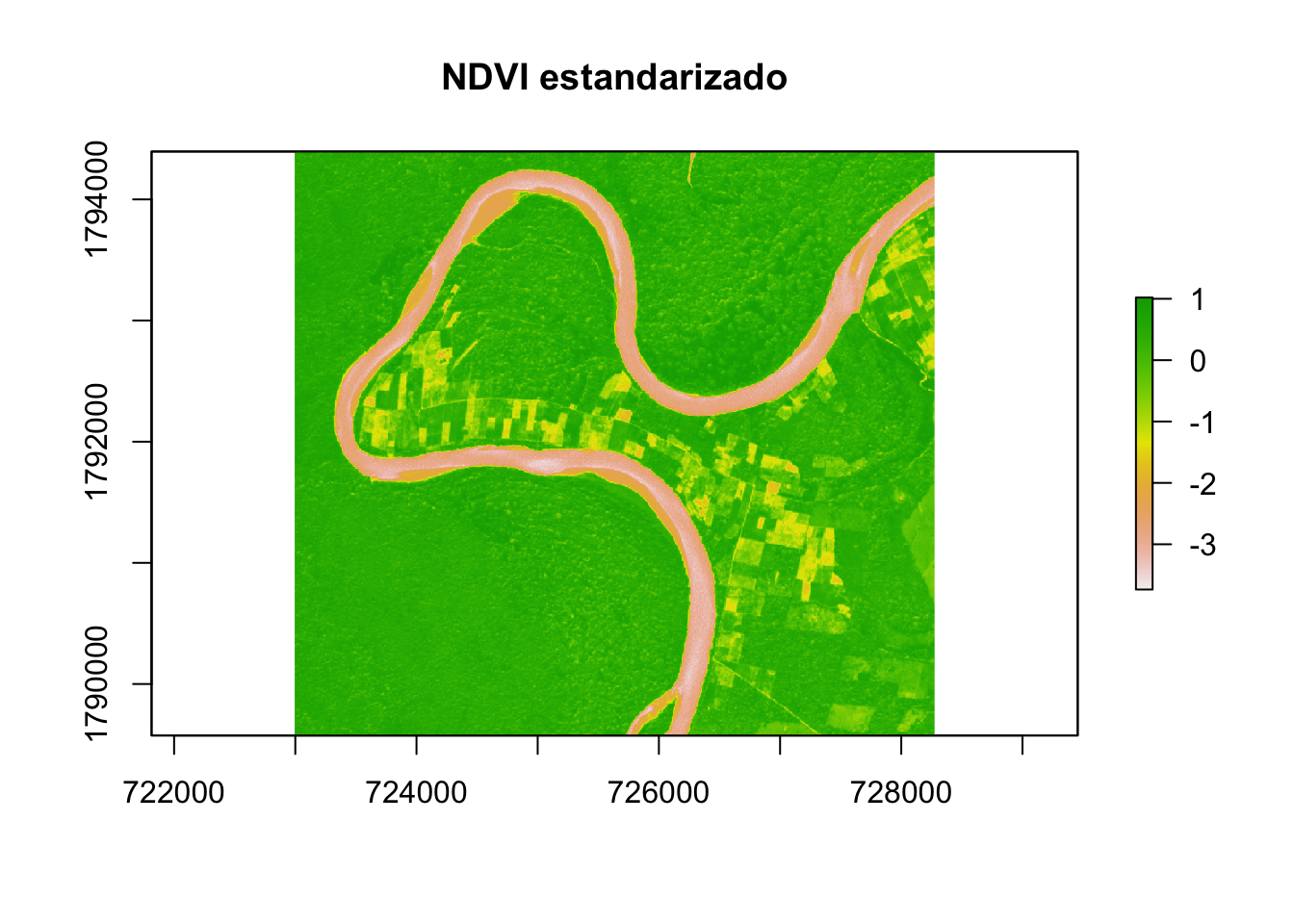
Con base en el procedimiento anterior, tratemos de generar un ejercicio simple de detección de cambios. Tomemos una imagen RapidEye del mismo lugar pero tomada un año después que la que acabamos de trabajar. Llevemos a cabo el mismo procedimiento para obtener su NDVI estandarizado.
chiapas2 <- brick("./data/2crop.tif")
VIS <- subset(chiapas2,subset=3)
NIR <- subset(chiapas2,subset=5)
chiapas2_ndvi <- (NIR-VIS)/(NIR+VIS)
chiapas2_ndvi_st <- (chiapas2_ndvi-cellStats(chiapas2_ndvi,stat="mean"))/(cellStats(chiapas2_ndvi,stat="sd"))
plot(chiapas2_ndvi_st,main="NDVI estandarizado, 1 año después")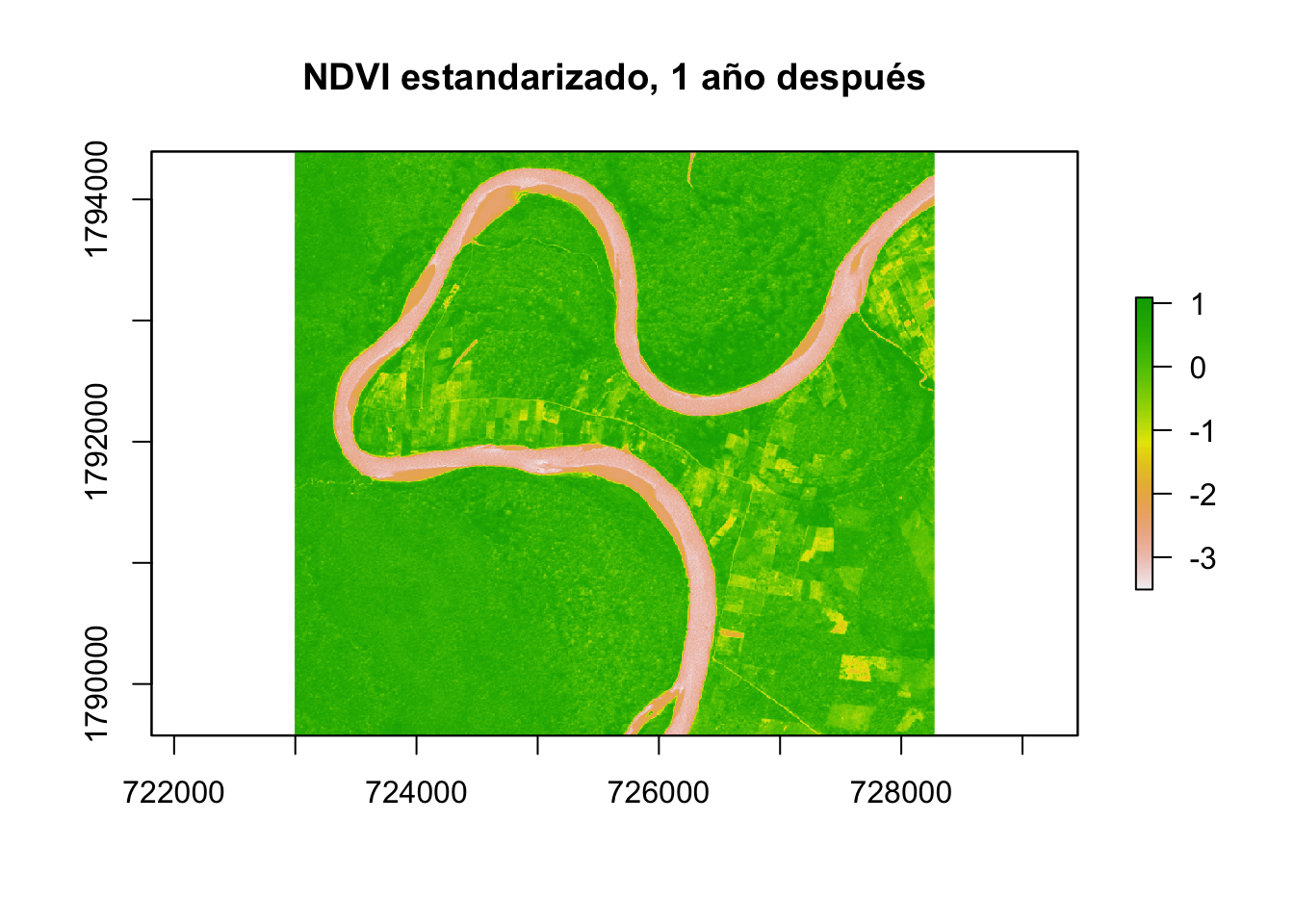
Ahora generaremos una imagen de diferencias, D, a partir de estas dos de NDVI.
\[ \begin{aligned} D = NDVI_1 - NDVI_2 \end{aligned} \]
D <- chiapas1_ndvi_st - chiapas2_ndvi_st
plot(D,main="Diferencias en NDVI")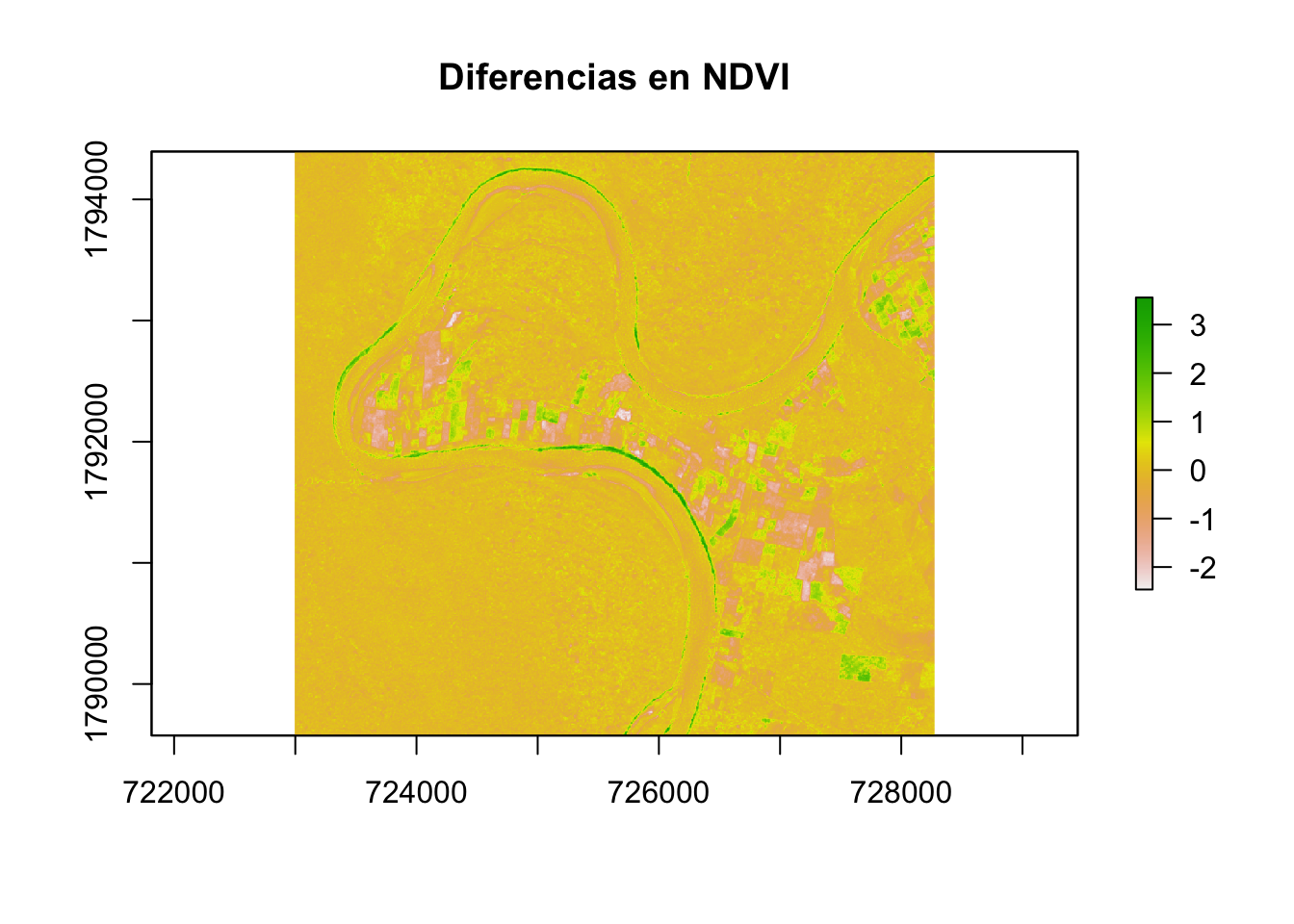
Aquí las diferencias positivas significan que el NDVI de la fecha 1 fue mayor al de la fecha 2. Diferencias negativas significan que el NDVI de la fecha 1 fue menor al de la fecha 2. Lo que dificulta el estudio de los cambios es que existe un continuo de magnitudes de diferencias. Naturalmente suponemos que diferencias de magnitud cercanas a \(0\) deben considerarse no-cambios pero no es claro de qué magnitud debe ser una diferencia para poder tomarse como un cambio. Vamos a visualizar el histograma de las diferencias para darnos una idea de la distribución de estas.
hist(D,breaks=100,main="Diferencias en NDVI",xlab="Diferencias",ylab="Conteos")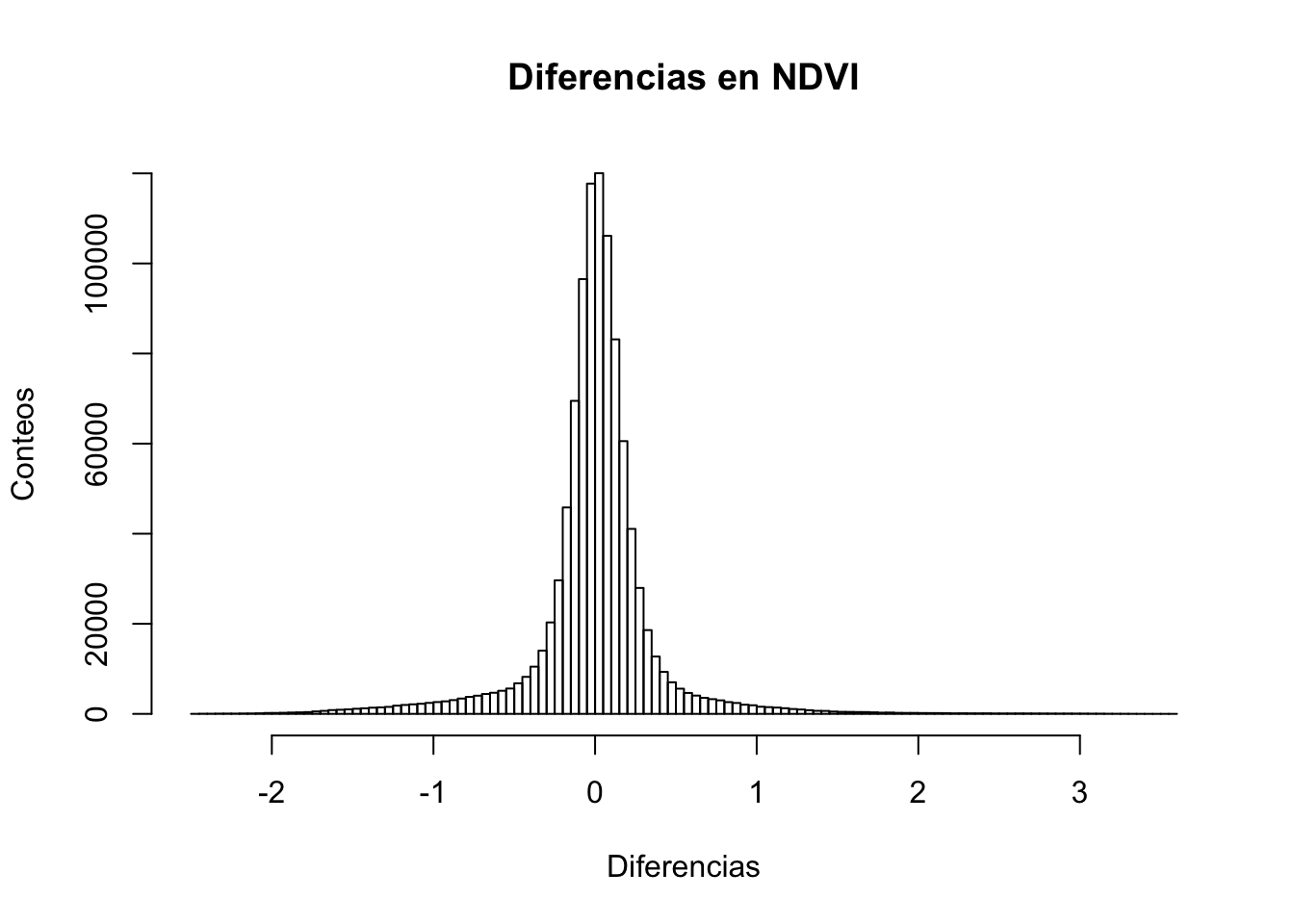
Parecería que la distribución de diferencias es aproximadamente normal. Se sabe que en una distribución normal, 95% de los valores se encuentran a 2 desviaciones estándar de su media. Tomando esto en cuenta definiremos como una diferencia significativa (cambio) a toda aquella que se encuentre a dos desviaciones estándar de la diferencia media. Podemos utilizar lo aprendido hasta ahora para condicionalmente reemplazar en la imagen de diferencias todo aquella que queremos descartar por ser una no-significativa.
umbral_positivo <- cellStats(D,stat="mean")+ 2*cellStats(D,stat="sd")
umbral_negativo <- cellStats(D,stat="mean")- 2*cellStats(D,stat="sd")
# Recordatorio: se pueden combinar múltiples condiciones utilizando operadores lógicos
#
# El operador | se usa para expresar "o"
#
# El operador & se usa para expresar "y"
D[(D>umbral_negativo) & (D<umbral_positivo)] <-NA Este es el resultado final de este proceso. En R, es tan secillo escribir un raster como leerlo. Para este propósito se utiliza la función writeRaster()
writeRaster(D, filename="./data/diferencias_significativas.tif", format="GTiff", overwrite=TRUE)
El paquete raster tiene funciones dedicadas a manejar la proyección de las capas con las que se está trabajando. Supongamos que queremos visualizar este último resultado en Google Earth. ¿está en la proyección correcta?
projection(D)## [1] "+proj=utm +zone=15 +datum=WGS84 +units=m +no_defs +ellps=WGS84 +towgs84=0,0,0"NO.
D_reproj <- projectRaster(D,crs=CRS("+proj=longlat"))Luego simplemente generamos un KML con la funcón KML()
KML(D_reproj,"./data/cambios_kml",maxpixels=1000000,overwrite=TRUE)13.3.5 Capas vectoriales, primeros pasos
Una Capa vectorial es una estructura espacial conformada por unidades (puntos, líneas o polígonos), asociada a una tabla de datos.
La librería GDAL es una librería Open Source para leer y escribir formatos raster y vectoriales. El paquete rgdal es una interfaz a esta librería que permite la fácil lectura de Shape files en R.
library("rgdal")## rgdal: version: 1.3-4, (SVN revision 766)
## Geospatial Data Abstraction Library extensions to R successfully loaded
## Loaded GDAL runtime: GDAL 2.1.3, released 2017/20/01
## Path to GDAL shared files: /Library/Frameworks/R.framework/Versions/3.5/Resources/library/rgdal/gdal
## GDAL binary built with GEOS: FALSE
## Loaded PROJ.4 runtime: Rel. 4.9.3, 15 August 2016, [PJ_VERSION: 493]
## Path to PROJ.4 shared files: /Library/Frameworks/R.framework/Versions/3.5/Resources/library/rgdal/proj
## Linking to sp version: 1.3-1puntos <- readOGR(dsn="./data/puntos.shp",layer="puntos")## OGR data source with driver: ESRI Shapefile
## Source: "/Users/jequihua/Documents/repositories/big-data-ambiental/data/puntos.shp", layer: "puntos"
## with 11 features
## It has 1 fields
## Integer64 fields read as strings: idlineas <- readOGR(dsn="./data/lineas.shp",layer="lineas")## OGR data source with driver: ESRI Shapefile
## Source: "/Users/jequihua/Documents/repositories/big-data-ambiental/data/lineas.shp", layer: "lineas"
## with 3 features
## It has 1 fields
## Integer64 fields read as strings: idpoligonos <- readOGR(dsn="./data/poligonos.shp",layer="poligonos")## OGR data source with driver: ESRI Shapefile
## Source: "/Users/jequihua/Documents/repositories/big-data-ambiental/data/poligonos.shp", layer: "poligonos"
## with 3 features
## It has 1 fields
## Integer64 fields read as strings: idpar(mfrow=c(1,3))
plot(puntos)
plot(lineas)
plot(poligonos)
Cargado un shape file en el espacio de trabajo de R, este se convierte en un data.frame espacial. Por lo que la mayoría de las funcionalidades disponibles para un data.frame aplican.
# tipo de objeto
class(puntos)## [1] "SpatialPointsDataFrame"
## attr(,"package")
## [1] "sp"class(lineas)## [1] "SpatialLinesDataFrame"
## attr(,"package")
## [1] "sp"class(poligonos)## [1] "SpatialPolygonsDataFrame"
## attr(,"package")
## [1] "sp"# cabeza de los datos
head(puntos)## id
## 1 11
## 2 10
## 3 9
## 4 8
## 5 7
## 6 6head(lineas)## id
## 0 3
## 1 2
## 2 1head(poligonos)## id
## 0 3
## 1 2
## 2 1Se debe notar que por como están programadas estas estructuras espaciales en R, todo supconjunto de un data.frame espacial es nuevamente un data.frame espacial y se pueden generar subconjuntos utilizando los mecanismos usuales para data.frames
# elegimos el primer polígono
poligono <- poligonos[1,]
class(poligono)## [1] "SpatialPolygonsDataFrame"
## attr(,"package")
## [1] "sp"plot(poligono)
Como sucede con un data.frame usual se puede agregar una nueva columna (variable) a un data.frame espacial utilizando el signo “$”.
# agregamos al objetos poligonos la variable intensidad. Este objeto tiene 3 polígonos. Asignarémos un valor de intensidad = 0 al primer polígono, y un valor de intensidad = 2 a los siguientes 2.
poligonos$intensidad <- c(1,2,2)
head(poligonos)## id intensidad
## 0 3 1
## 1 2 2
## 2 1 2colores <- c("red","blue")
plot(poligonos,col=colores[poligonos$intensidad])
13.3.6 Análisis de capas vectoriales usando R
Existen numerosas librerías para hacer análisis de capas vectoriales de puntos, líneas y polígonos.
Algo que es importante es entender cómo manipular distintas capas vectoriales simultáneamente, por lo que vale la pena leer las funciones disponibles en los paquetes más fundamentales: sp, maptools, raster, etc.
Por ejemplo, es trivial encontrar las intersecciones espaciales entre polígonos y puntos utilizando la función over() del paquete sp.
overlay <- over(puntos,poligonos)
overlay## id intensidad
## 1 3 1
## 2 3 1
## 3 3 1
## 4 3 1
## 5 3 1
## 6 3 1
## 7 3 1
## 8 3 1
## 9 3 1
## 10 2 2
## 11 1 2plot(poligonos,col=colores[poligonos$intensidad])
plot(puntos,add=TRUE)
La función over() está programada para manejar de manera distinta cada una de los casos que surgen de las combinaciones de puntos, líneas y polígonos. Es recomendable consultar la ayuda de la función para entender cada caso (?over). En el caso de puntos vs polígonos la salida tiene tantas filas como puntos hay de entrada (11). Para cada fila de la salida se indica sobre qué polígono cae cada punto y cualquier otra variable que esté incluída en la tabla de datos de los polígonos (e.g. intensidad en este caso.)
Para determinar intersecciones complejas entre dos capas de polígonos es más conveniente utilizar el paquete raster.
poligonos2 <- readOGR(dsn="./data/cruces.shp",layer="cruces")## OGR data source with driver: ESRI Shapefile
## Source: "/Users/jequihua/Documents/repositories/big-data-ambiental/data/cruces.shp", layer: "cruces"
## with 4 features
## It has 1 fields
## Integer64 fields read as strings: idplot(poligonos2)
plot(poligonos,col=colores[poligonos$intensidad],add=TRUE)
intersecciones <- poligonos + poligonos2## Loading required namespace: rgeosdata.frame(intersecciones)## id.1 intensidad id.2
## 1 3 1 <NA>
## 2 2 2 <NA>
## 3 1 2 <NA>
## 4 <NA> NA 4
## 5 <NA> NA 3
## 6 <NA> NA 2
## 7 <NA> NA 1
## 8 3 1 4
## 9 3 1 3
## 10 3 1 2
## 11 3 1 1
## 12 2 2 3
## 13 1 2 2
## 14 1 2 1plot(intersecciones,col=blues9)
13.3.7 Juntando todo
La función extract() del paquete raster es una sumamente útil. Permite sobreponer un data.frame espacial sobre un raster y extraer los valores de píxeles sobre los que cae cada registro del data.frame espacial.
Cuando el data.frame espacial es uno de puntos entonces cada punto cae exáctamente sobre 1 pixel por lo que la extracción es trivial.
Si el data.frame espacial es, por ejemplo, uno de polígonos; entonces cada registro cae sobre un grupo de píxeles. En este caso, la función extract debe recibir una función para poder asociarle un valor a cada polígono. Por ejemplo, si se elige la funciòn promedio, cada polígono es asociado al promedio de píxeles que envuelve.
# leamos un raster de altitud con resolución de 1 km
altitud <- raster("./data/dem30_mean1000.tif")
plot(altitud)
plot(poligonos,col=colores[poligonos$intensidad],add=TRUE)
plot(puntos,add=TRUE)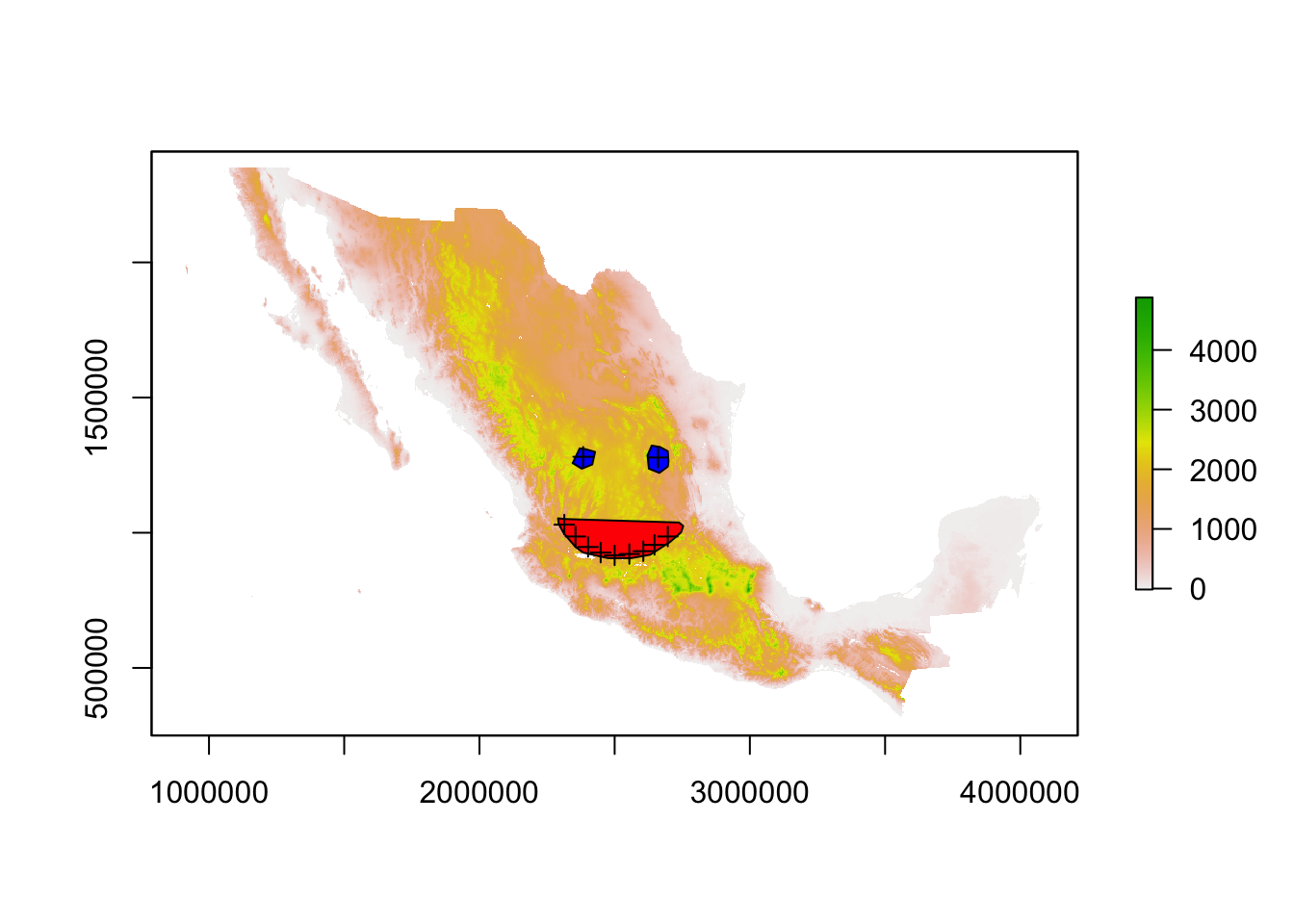
extraccion_puntos <- extract(altitud,puntos)
# valor de altitud de cada punto
extraccion_puntos## [1] 2152.546 1914.298 1723.259 1756.557 1732.879 1531.412 1860.138
## [8] 1442.991 2103.941 1707.098 2133.286extraccion_poligonos <- extract(altitud,poligonos,fun=mean,na.rm=TRUE)
# valor de altitud promedio por polígono
extraccion_poligonos## [,1]
## [1,] 1866.597
## [2,] 1682.829
## [3,] 2140.274Podemos combinar múltiples funciones para lograr objetivos muy específicos. Por ejemplo, supongamos que queremos cortar el raster de altitud con base en nuestro shape de polígonos.
extShape <- extent(poligonos)
img <- crop(altitud, extShape, snap='out')
img.NA <- setValues(img, NA)
img.mask <- rasterize(poligonos, img.NA)
img.crop <- mask(x=img, mask=img.mask)
plot(img.crop,main="Altitud en polígonos")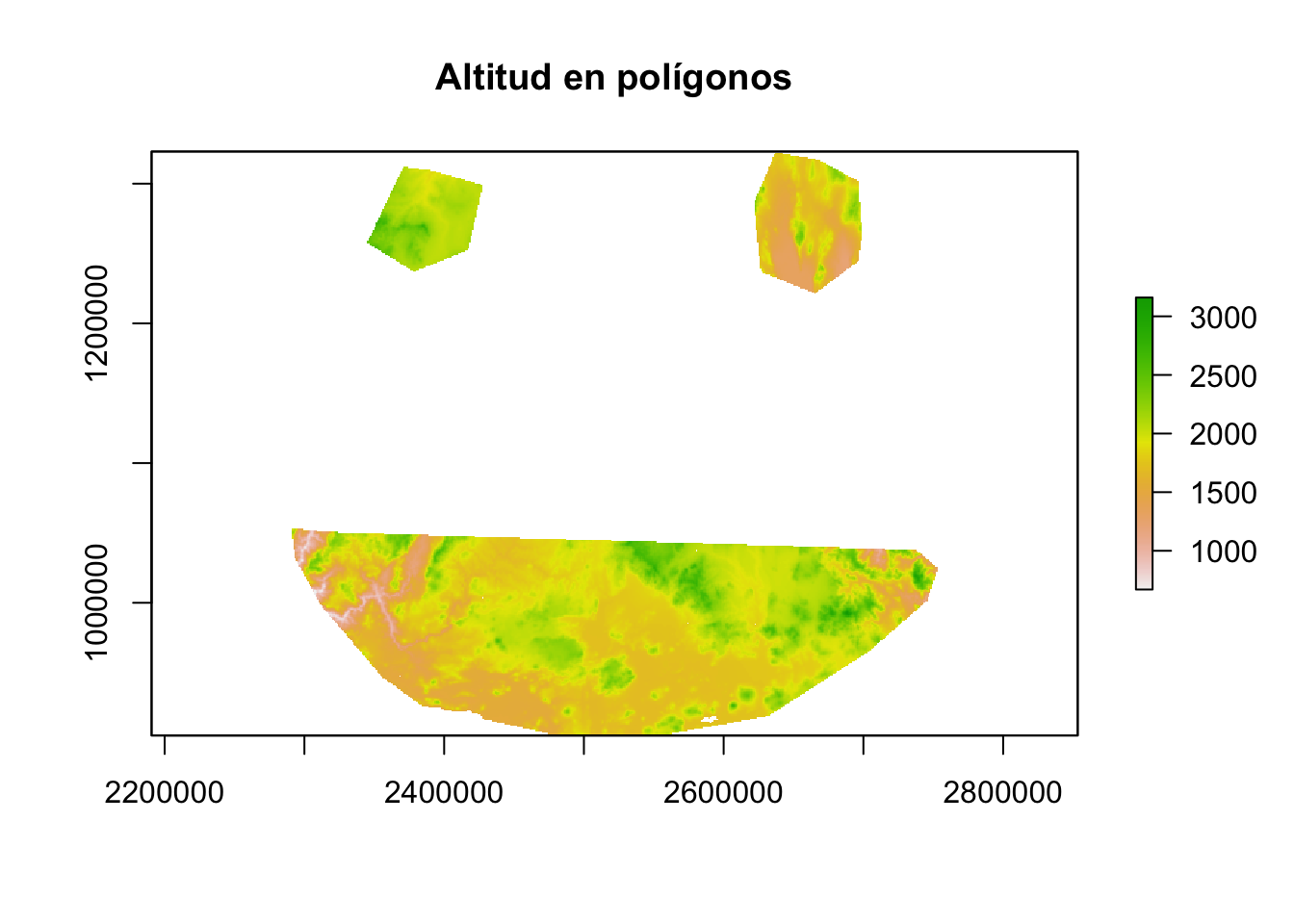
Ahora llevaremos a cabo un ejercicio en el que usaremos varias distintas funcionalidades de R para generar una regionalización del país de manera automatizada.
Tomemos el raster altitud como base.
Vamos a inicializar un raster multiespectral vacío y luego asignarle el raster de altitud como primera banda.
# raster multiespectral vacío
brik <- brick()
# asignemos altitud como primera banda
brik <- addLayer(brik,altitud)El raster multiespectral brik, hereda los metadatos de el raster altitud. Si queremos introducirle más bandas, estas tienen que compartir exactamente los mismos metadatos. Introduzcamos las variables % vegetacion boscosa, % vegetación no-boscosa, % de no-vegetación, temperatura máxima promedio, temperatura mínima promedio y precipitación promedio.
v_boscosa <- raster("./data/MOD44B_2014-03-06.Percent_Tree_Cover.tif")
v_noboscosa <- raster("./data/MOD44B_2014-03-06.Percent_NonTree_Vegetation.tif")
v_no <- raster("./data/MOD44B_2014-03-06.Percent_NonVegetated.tif")
# plot v_boscosa
plot(v_boscosa,main="% vegetación boscosa")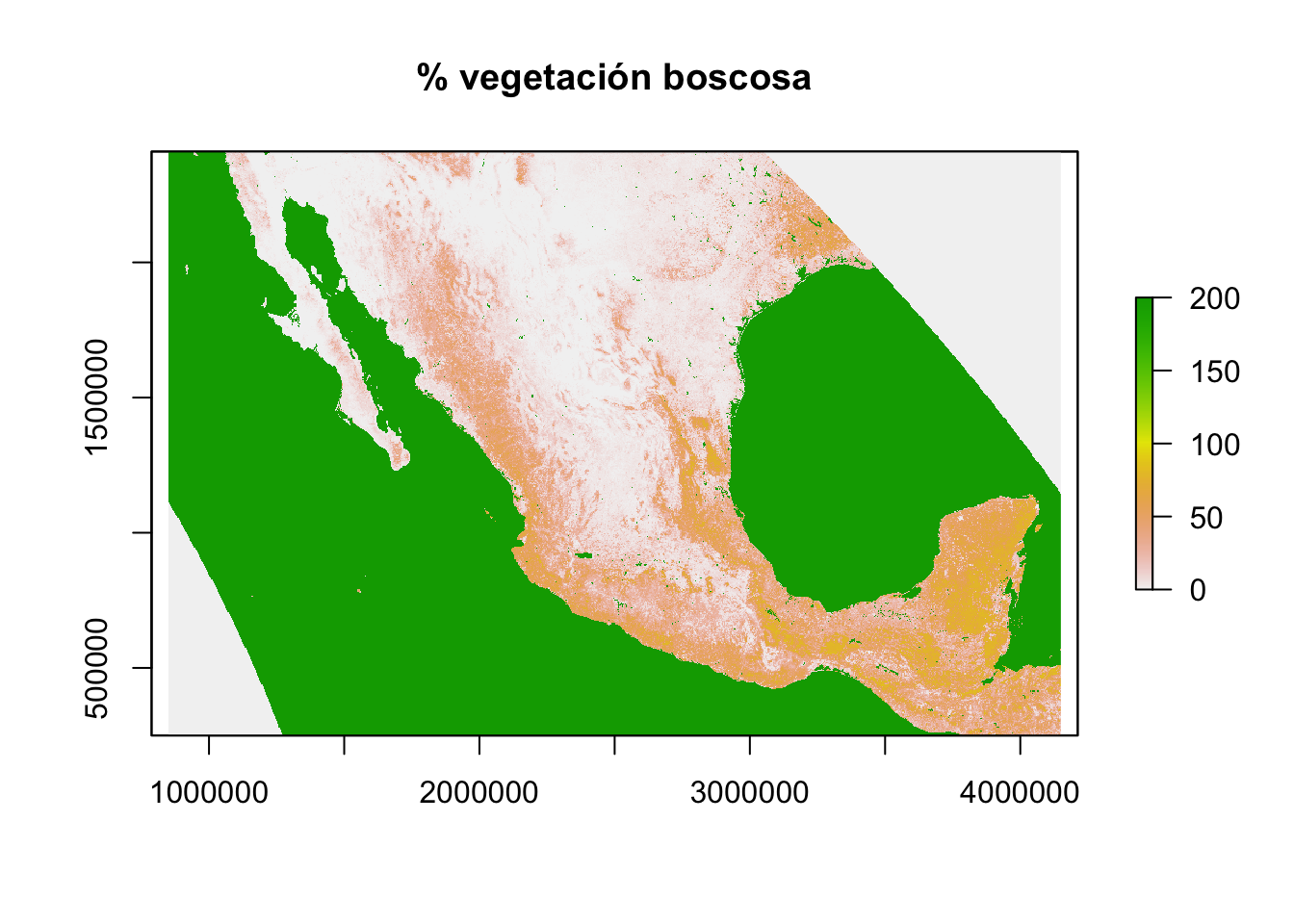
# ¿hay un match entre los metadatos de estas capas y el raster altitud?
projection(v_boscosa)==projection(altitud)## [1] TRUEextent(v_boscosa)==extent(altitud)## [1] TRUEres(v_boscosa)==res(altitud)## [1] FALSE FALSE# resolución de v_boscosa?
res(v_boscosa)## [1] 250 250Todo está harmonizado excepto la resolución de los rasters. Las tres capas de vegetación anteriores están generadas con base en imágenes modis de 250 m. Sabemos que nuestro raster de altitud tiene una resolución de 1000 m. Agreguemos estas capas por un factor de 4 con base en la función promedio. También observamos que estas capas se extienden más allá de la delimitación oficial de México. Usaremos el raster altitud para eliminar todo aquello que no corresponda con México. Luego podremos agregar las capas de vegetación como bandas nuevas a nuestro raster multiespectral.
# agregación factor *4 con base en función promedio
v_boscosa <- aggregate(v_boscosa,fact=4,fun=mean,na.rm=TRUE)
v_noboscosa <- aggregate(v_noboscosa,fact=4,fun=mean,na.rm=TRUE)
v_no <- aggregate(v_no,fact=4,fun=mean,na.rm=TRUE)
# filtrado de capas: si es NA en altitud que sea NA en capas de vegetación
v_boscosa[is.na(altitud)]<-NA
v_noboscosa[is.na(altitud)]<-NA
v_no[is.na(altitud)]<-NA
# Introduzcamos estas capas a nuestro raster multiespectral
brik <- addLayer(brik,v_boscosa)
brik <- addLayer(brik,v_noboscosa)
brik <- addLayer(brik,v_no)
dim(brik)## [1] 2160 3300 4Ahora simplemente agregaremos las capas que faltan al raster multiespectral: temperatura máxima promedio, temperatura mínima promedio y precipitación promedio
# Agregamos las bandas que faltan
brik <- addLayer(brik,
raster("./data/temp_max.tif"))
brik <- addLayer(brik,
raster("./data/temp_min.tif"))
brik <- addLayer(brik,
raster("./data/precipitacion.tif"))Empezamos discutiendo matrices y data.frames. Los data.frames son estructuras sumamente apropiadas para llevar a cabo análisis. Vamos a generar un data.frame a partir de nuestro raster multiespectral. Cada fila correspondrá a un pixel. Cada banda se hará una columna (variable), además de las coordenadas x, y.
# tabla de datos
tabla_datos <- rasterToPoints(brik)
# descartemos las filas no completas
tabla_datos_clean <- tabla_datos[complete.cases(tabla_datos),]
head(tabla_datos_clean)## x y dem30_mean1000 MOD44B_2014.03.06.Percent_Tree_Cover
## [1,] 1076500 2349500 110.13097 1.3125
## [2,] 1077500 2349500 65.34465 1.1875
## [3,] 1078500 2349500 51.05413 1.1875
## [4,] 1085500 2349500 153.33623 1.3125
## [5,] 1086500 2349500 158.40757 1.3750
## [6,] 1087500 2349500 161.25201 0.0000
## MOD44B_2014.03.06.Percent_NonTree_Vegetation
## [1,] 23.3750
## [2,] 32.8750
## [3,] 37.1250
## [4,] 28.8750
## [5,] 38.1875
## [6,] 10.7500
## MOD44B_2014.03.06.Percent_NonVegetated temp_max temp_min
## [1,] 75.3125 21.62551 11.85019
## [2,] 65.9375 21.84063 12.31568
## [3,] 61.6875 22.09170 12.62078
## [4,] 69.8125 22.64403 11.68126
## [5,] 60.4375 22.73465 11.69161
## [6,] 89.2500 22.78917 11.58576
## precipitacion
## [1,] 23.25161
## [2,] 20.96546
## [3,] 19.42261
## [4,] 22.58016
## [5,] 22.40759
## [6,] 22.75284Es evidente que estas variables deben estar correlacionadas. Generemos un análisis de componentes principales sobre estas variables sin tomar en cuenta las coordenadas.
# cálculo de componentes principales
componentes <- prcomp(tabla_datos_clean[,2:9],center=TRUE,scale. =TRUE)
# varianza explicada
summary(componentes)## Importance of components:
## PC1 PC2 PC3 PC4 PC5 PC6
## Standard deviation 1.9101 1.4038 1.0478 0.64277 0.63047 0.54975
## Proportion of Variance 0.4561 0.2463 0.1373 0.05164 0.04969 0.03778
## Cumulative Proportion 0.4561 0.7024 0.8396 0.89128 0.94097 0.97874
## PC7 PC8
## Standard deviation 0.34626 0.22396
## Proportion of Variance 0.01499 0.00627
## Cumulative Proportion 0.99373 1.00000Ahora utilicemos 4 componentes principales para llevar a cabo un ejercicio de clustering usando k-medias
# k-medias, k=10
kmedias <- kmeans(componentes$x[,1:4],centers=10,iter.max=50)## Warning: Quick-TRANSfer stage steps exceeded maximum (= 96668250)El resultado del clustering se lo podemos asociar a las coordenadas de nuestra matriz inicial. Luego convertir esta tabla en un objeto espacial y luego un raster.
# clustering result
clustered_data <- data.frame(x=tabla_datos_clean[,1],
y=tabla_datos_clean[,2],
clust=kmedias$cluster)
# convertir esta tabla en un objeto espacial
coordinates(clustered_data)=~x+y
gridded(clustered_data)=TRUE## Warning in points2grid(points, tolerance, round): grid has empty column/
## rows in dimension 1clustered_data<-raster(clustered_data)
projection(clustered_data)<-projection(altitud)Finalmente podemos convertir este raster en una capa vectorial y guardarla en el disco duro.
#poligonos_clusters <- rasterToPolygons(clustered_data,n=4,dissolve=TRUE)
#writeOGR(poligonos_clusters,"./data/regionalizacion.shp",
# "regionalizacion",driver="ESRI Shapefile")