8.3 “Aprendizaje de parámetros” o “estructuración de la red”
El comando bn.fit genera estimaciones de parámetros para las tablas de probabilidad condicionales en cada nodo. Sin embargo, el comando bn.fit requiere que la estructura de red represente un DAG (gráfico acíclico dirigido), “de lo contrario no se pueden estimar sus parámetros porque la factorización de la distribución de probabilidad global de los datos en los locales (uno para cada variable en el modelo) no se conoce completamente”. Por lo tanto, los arcos no dirigidos deben establecerse antes de la estimación de parámetros. Vemos en la estructura anterior estimada que el nodo “A” no está conectado a la estructura de red. Por lo tanto, antes de aplicar bn.fit, debemos establecer un arco direccional para A. debido a la opinión “experta”, el conocimiento del sistema, o estudios previos, vamos a establecer un arco entre A y T. El método predeterminado para la estimación de parámetros es el de máxima verosimilitud (MLE).
#creating a new DAG with the A to T relationship(based on our previous knowledge that goint to asia effects having tuberculosis)
hcex1 = set.arc(hcex, from = "A", to = "T")
plot(hcex1)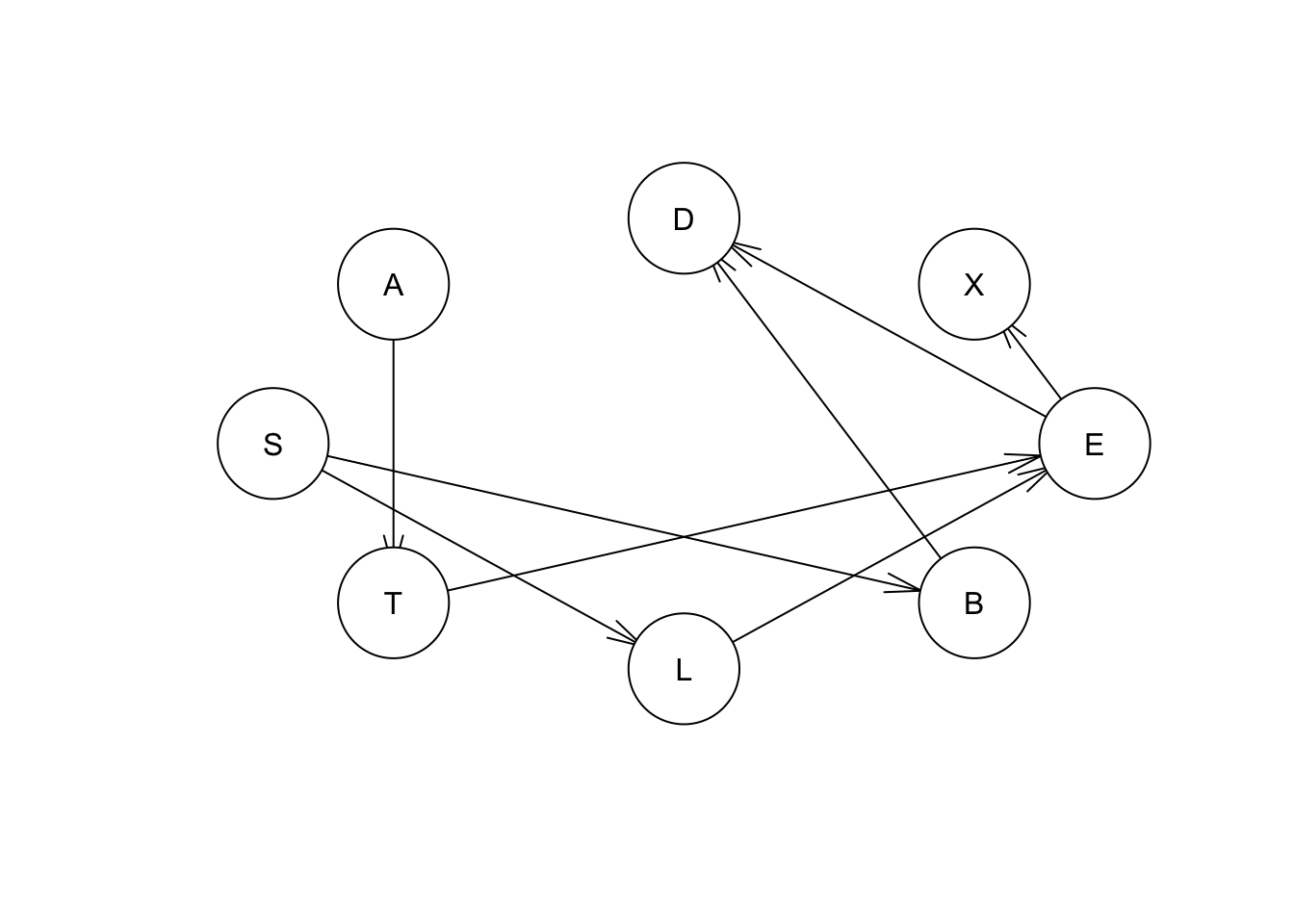
Ahora podemos ejecutar el comando bn.fit para obtener los estimadores de los parámetros:
# fitting the network with conditoinal probability tables
fit = bn.fit(hcex1, asia)
fit ##
## Bayesian network parameters
##
## Parameters of node A (multinomial distribution)
##
## Conditional probability table:
## no yes
## 0.9916 0.0084
##
## Parameters of node S (multinomial distribution)
##
## Conditional probability table:
## no yes
## 0.497 0.503
##
## Parameters of node T (multinomial distribution)
##
## Conditional probability table:
##
## A
## T no yes
## no 0.991528842 0.952380952
## yes 0.008471158 0.047619048
##
## Parameters of node L (multinomial distribution)
##
## Conditional probability table:
##
## S
## L no yes
## no 0.98631791 0.88230616
## yes 0.01368209 0.11769384
##
## Parameters of node B (multinomial distribution)
##
## Conditional probability table:
##
## S
## B no yes
## no 0.7006036 0.2823062
## yes 0.2993964 0.7176938
##
## Parameters of node E (multinomial distribution)
##
## Conditional probability table:
##
## , , L = no
##
## T
## E no yes
## no 1 0
## yes 0 1
##
## , , L = yes
##
## T
## E no yes
## no 0 0
## yes 1 1
##
##
## Parameters of node X (multinomial distribution)
##
## Conditional probability table:
##
## E
## X no yes
## no 0.956587473 0.005405405
## yes 0.043412527 0.994594595
##
## Parameters of node D (multinomial distribution)
##
## Conditional probability table:
##
## , , E = no
##
## B
## D no yes
## no 0.90017286 0.21373057
## yes 0.09982714 0.78626943
##
## , , E = yes
##
## B
## D no yes
## no 0.27737226 0.14592275
## yes 0.72262774 0.85407725Podemos recuperar la probabilidad condicional de un nodo específico mediante el operador usual “$”:
fit$L##
## Parameters of node L (multinomial distribution)
##
## Conditional probability table:
##
## S
## L no yes
## no 0.98631791 0.88230616
## yes 0.01368209 0.11769384fit$D##
## Parameters of node D (multinomial distribution)
##
## Conditional probability table:
##
## , , E = no
##
## B
## D no yes
## no 0.90017286 0.21373057
## yes 0.09982714 0.78626943
##
## , , E = yes
##
## B
## D no yes
## no 0.27737226 0.14592275
## yes 0.72262774 0.85407725Si lo queremos, podemos también visualizar la tabla de probabilidad condicional mmediante gráficas de barras:
bn.fit.barchart(fit$D)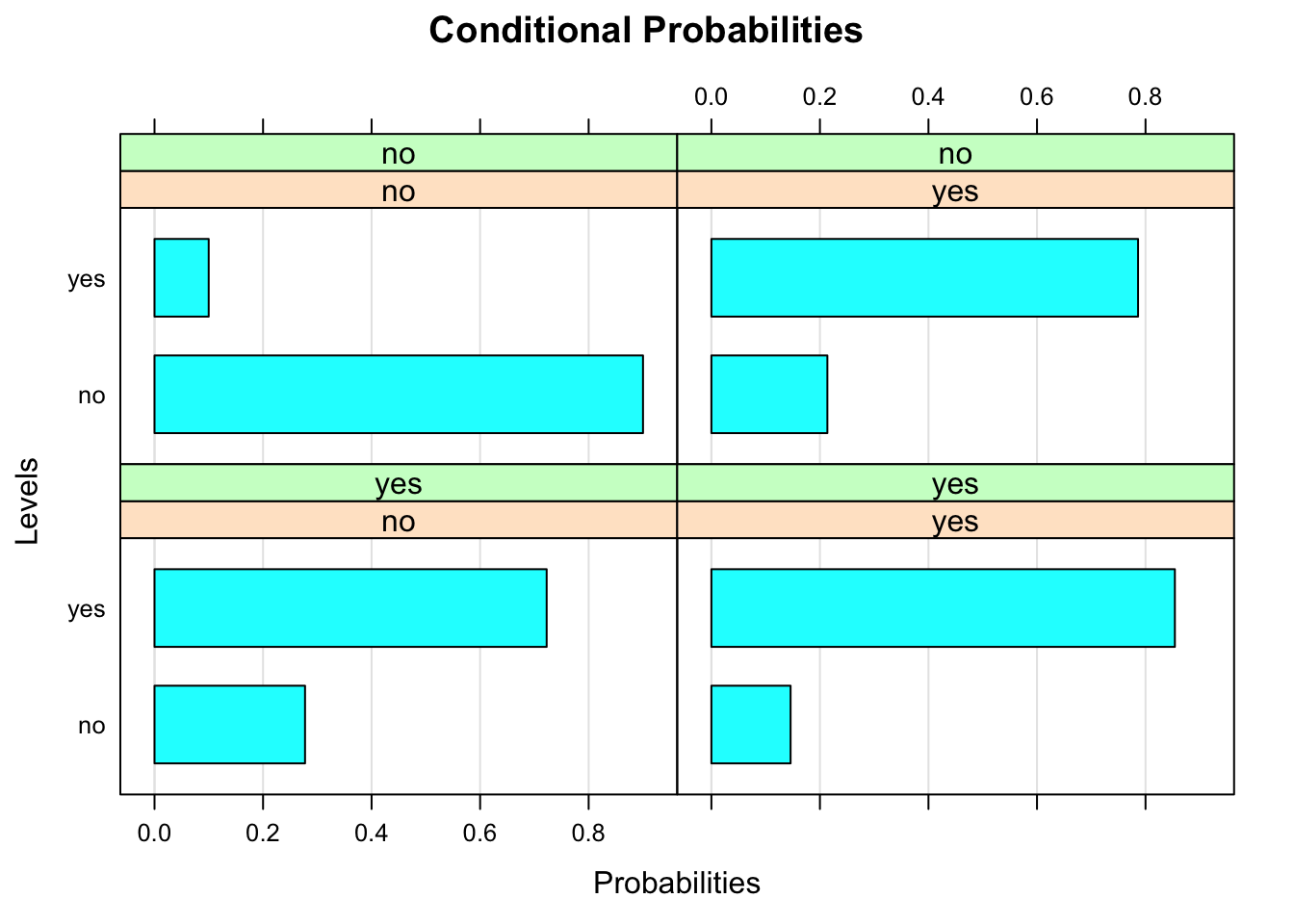
o, si lo preferimos, como una gráfica de puntos:
bn.fit.dotplot(fit$D)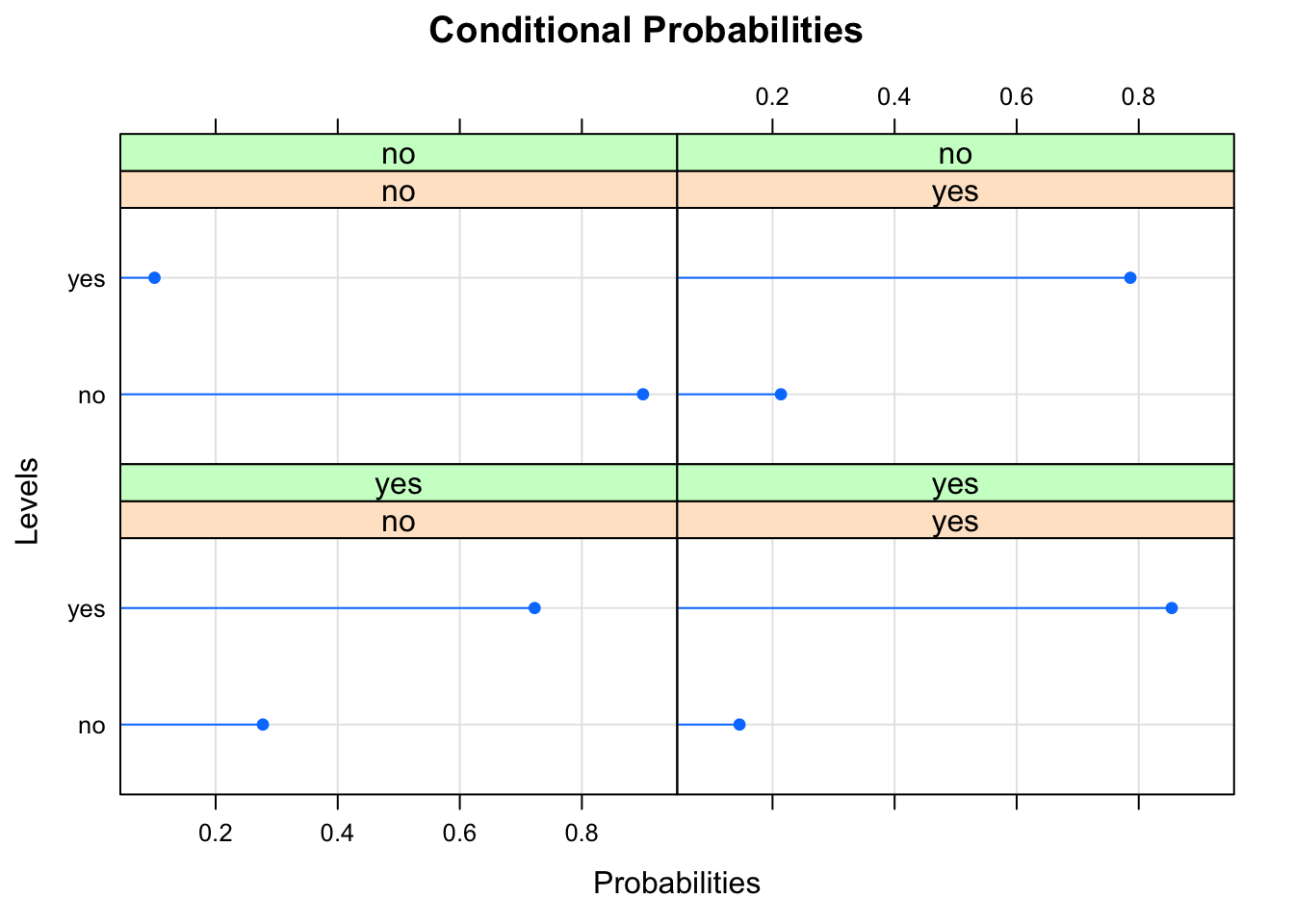
In addition to maximum likelihood, parameter estimation can be done performed with Bayesian methods - but currently only with discrete data. Below is an example with the Asia dataset. The same bn.fit command line is used, but the method is specified as ‘bayes’.
fit1 = bn.fit(hcex1, asia, method = "bayes")
fit1##
## Bayesian network parameters
##
## Parameters of node A (multinomial distribution)
##
## Conditional probability table:
## no yes
## 0.9915017 0.0084983
##
## Parameters of node S (multinomial distribution)
##
## Conditional probability table:
## no yes
## 0.4970006 0.5029994
##
## Parameters of node T (multinomial distribution)
##
## Conditional probability table:
##
## A
## T no yes
## no 0.991479278 0.947058824
## yes 0.008520722 0.052941176
##
## Parameters of node L (multinomial distribution)
##
## Conditional probability table:
##
## S
## L no yes
## no 0.98622008 0.88223017
## yes 0.01377992 0.11776983
##
## Parameters of node B (multinomial distribution)
##
## Conditional probability table:
##
## S
## B no yes
## no 0.7005633 0.2823494
## yes 0.2994367 0.7176506
##
## Parameters of node E (multinomial distribution)
##
## Conditional probability table:
##
## , , L = no
##
## T
## E no yes
## no 9.999730e-01 3.105590e-03
## yes 2.699638e-05 9.968944e-01
##
## , , L = yes
##
## T
## E no yes
## no 3.831418e-04 2.941176e-02
## yes 9.996169e-01 9.705882e-01
##
##
## Parameters of node X (multinomial distribution)
##
## Conditional probability table:
##
## E
## X no yes
## no 0.956538171 0.006072874
## yes 0.043461829 0.993927126
##
## Parameters of node D (multinomial distribution)
##
## Conditional probability table:
##
## , , E = no
##
## B
## D no yes
## no 0.90012963 0.21376147
## yes 0.09987037 0.78623853
##
## , , E = yes
##
## B
## D no yes
## no 0.27777778 0.14630225
## yes 0.72222222 0.85369775